Japanese
English
- 有料閲覧
- Abstract 文献概要
- 1ページ目 Look Inside
- 参考文献 Reference
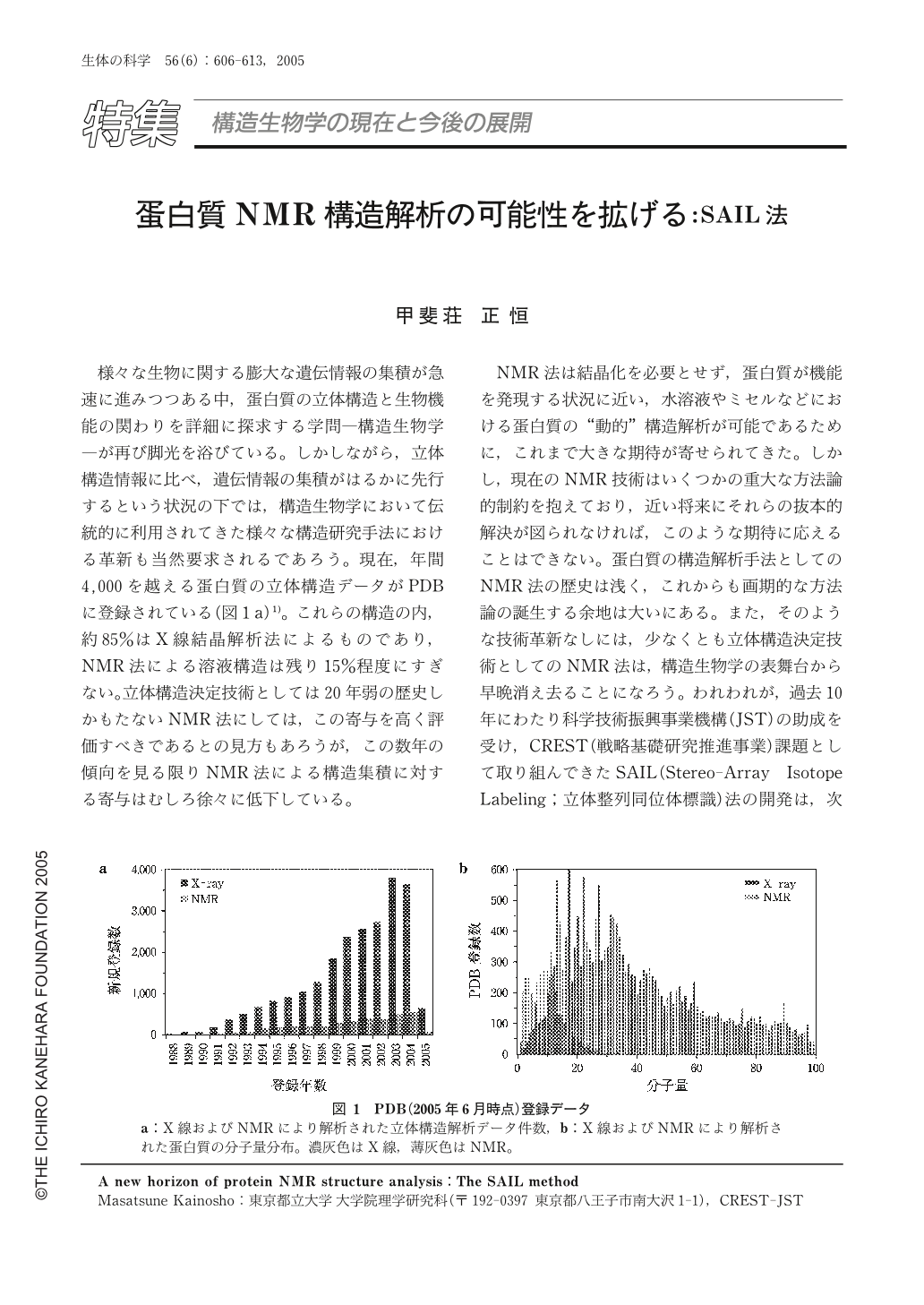
様々な生物に関する膨大な遺伝情報の集積が急速に進みつつある中,蛋白質の立体構造と生物機能の関わりを詳細に探求する学問―構造生物学―が再び脚光を浴びている。しかしながら,立体構造情報に比べ,遺伝情報の集積がはるかに先行するという状況の下では,構造生物学において伝統的に利用されてきた様々な構造研究手法における革新も当然要求されるであろう。現在,年間4,000を越える蛋白質の立体構造データがPDBに登録されている(図1a)1)。これらの構造の内,約85%はX線結晶解析法によるものであり,NMR法による溶液構造は残り15%程度にすぎない。立体構造決定技術としては20年弱の歴史しかもたないNMR法にしては,この寄与を高く評価すべきであるとの見方もあろうが,この数年の傾向を見る限りNMR法による構造集積に対する寄与はむしろ徐々に低下している。
NMR法は結晶化を必要とせず,蛋白質が機能を発現する状況に近い,水溶液やミセルなどにおける蛋白質の“動的”構造解析が可能であるために,これまで大きな期待が寄せられてきた。しかし,現在のNMR技術はいくつかの重大な方法論的制約を抱えており,近い将来にそれらの抜本的解決が図られなければ,このような期待に応えることはできない。蛋白質の構造解析手法としてのNMR法の歴史は浅く,これからも画期的な方法論の誕生する余地は大いにある。また,そのような技術革新なしには,少なくとも立体構造決定技術としてのNMR法は,構造生物学の表舞台から早晩消え去ることになろう。われわれが,過去10年にわたり科学技術振興事業機構(JST)の助成を受け,CREST(戦略基礎研究推進事業)課題として取り組んできたSAIL(Stereo-Array Isotope Labeling;立体整列同位体標識)法の開発は,次世代の蛋白質NMR解析技術の発展へ向けて,新たな基盤を与えるものである2-5)。
Copyright © 2005, THE ICHIRO KANEHARA FOUNDATION. All rights reserved.


